母比率の差の検定をExploratoryで行う
この記事では、Exploratoryで母比率の差の検定を行ってみます。
例として、2つの患者の母集団があって、それぞれから取り出したサンプルサイズ(patients列)の内、何人が喫煙者か(smokers列)のデータがあるとします。
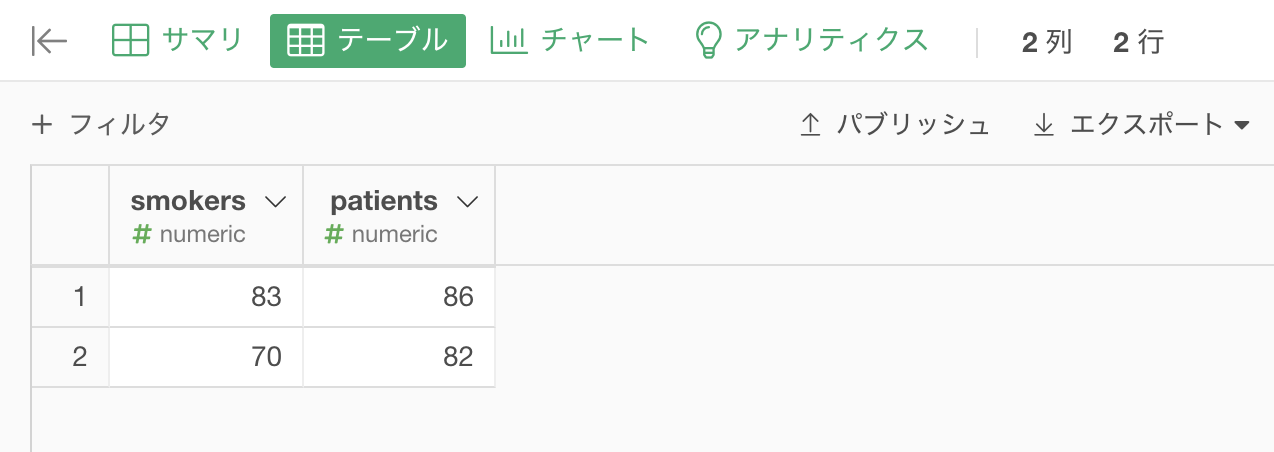
このサンプルデータを元に、これらの2つの母集団において、喫煙者の割合に差があるといえるのかどうかを母比率の差の検定で検定してみます。
カスタムRコマンドのステップを追加して、母比率の差の検定のモデルを、データフレームの列として作成します。プラス(+)アイコンをクリックして現れるメニューから、カスタムRコマンドを選びます。
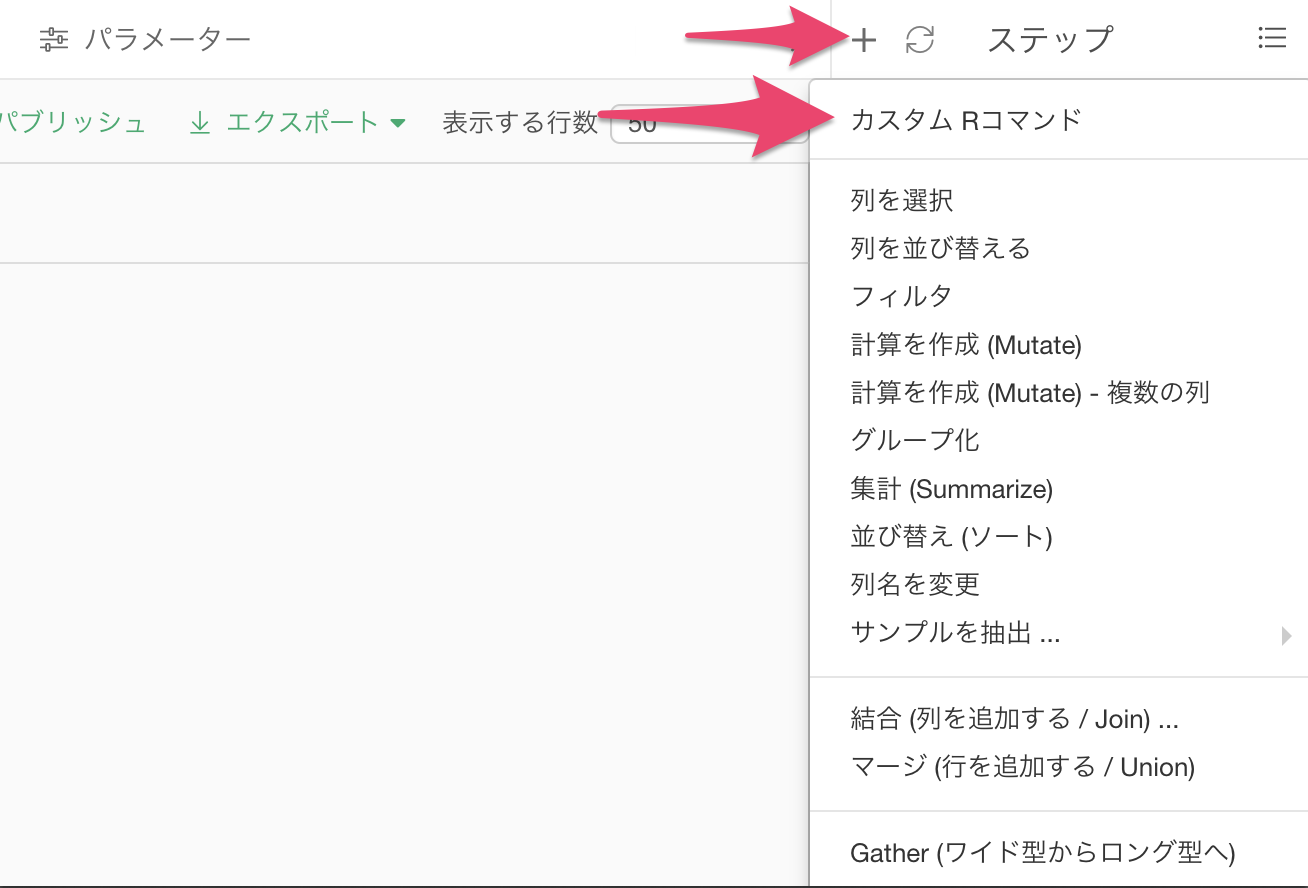
カスタムRコマンドダイアログが現れます。以下のRコマンドを入力します。
do(model=prop.test(.$smokers, .$patients))
.は元のデータフレームを表し、.smokers、.patientsはそれぞれ、このデータフレームのsmokers列、patients列を表します。 prop.test()は母比率の差の検定を行うRの関数で、この関数が作成したモデルをmodel列に格納する、というのがこのコマンドの行っている内容です。
実行ボタンをクリックすると、以下のように、model列を持ったデータフレームのステップが生成されます。
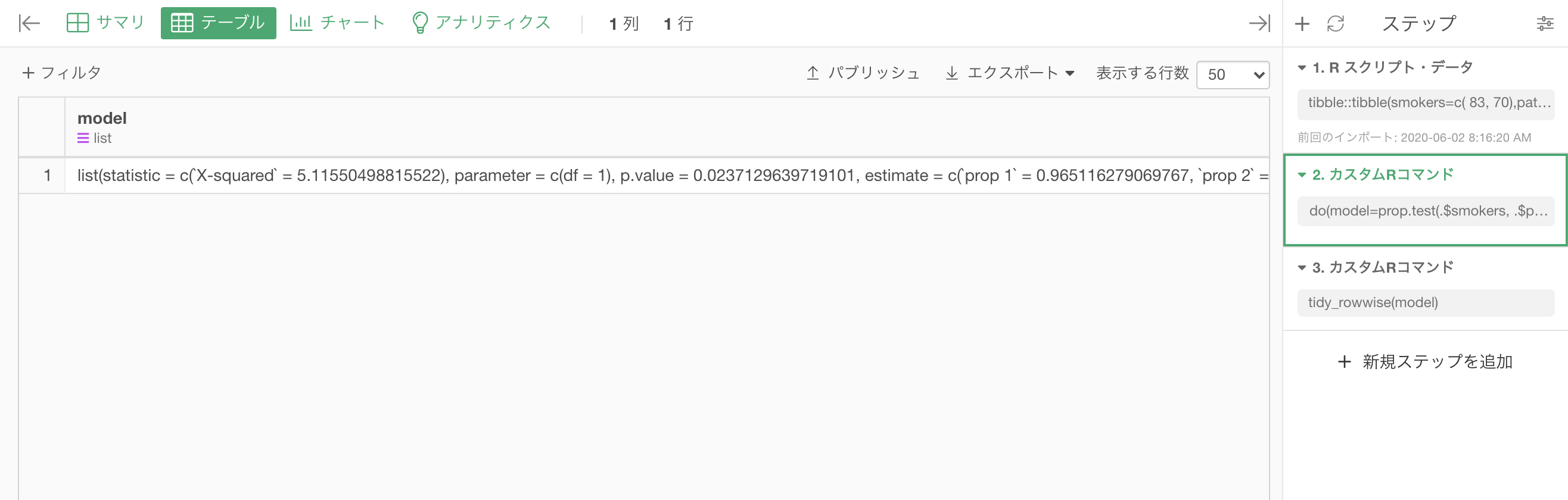
テスト結果を抽出するには、以下のコマンドを実行するカスタムステップを追加します。
model_info(model, output="summary")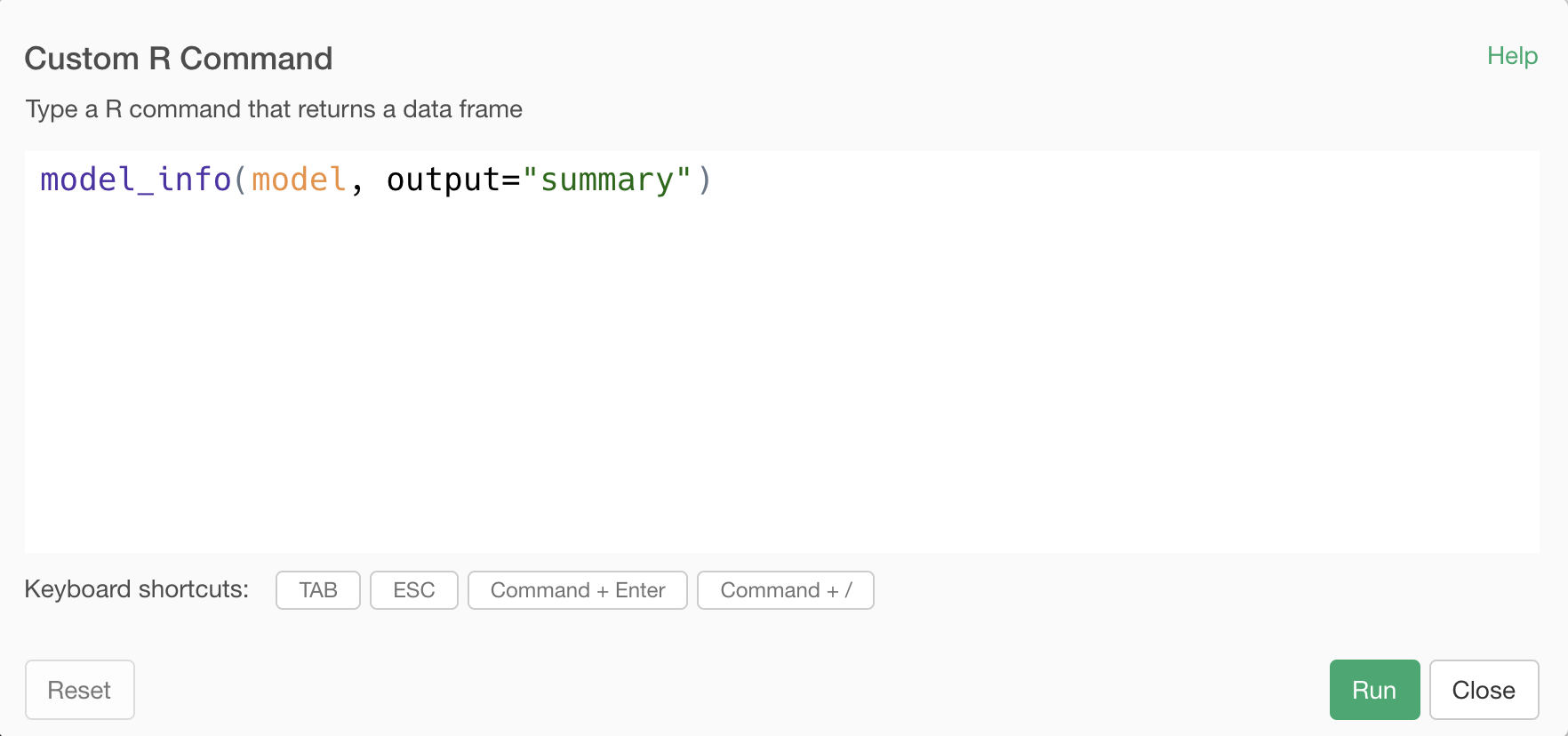
model_infoは、BroomというRパッケージの関数(この場合はglanceという関数)をデータフレームの列に格納されたモデルに対して呼び出すためのExploratoryの関数です。Broomパッケージは、統計・機械学習モデルを、tidyな形のデータフレームとして取り扱うための便利な関数のセットを提供しています。今回使用するglance関数は、モデルの情報を抜き出してデータフレームにするものです。
このステップが実行されると、テスト結果のサマリ情報を持ったステップが生成されます。

この場合、P値(p.value列)は0.0237となり、0.05を基準とするのであれば、2つの患者の母集団の間には、喫煙者の割合に有意な差があるといえるレベルだと言えます。